Перейти к:
Генетическая архитектура семейной гиперхолестеринемии на примере когорты жителей Санкт-Петербурга
https://doi.org/10.15829/1560-4071-2025-6432
EDN: MCOSAZ
Аннотация
Цель. Определение частоты патогенных и вероятно патогенных вариантов, ассоциированных с семейной гиперхолестеринемией (СГХС), с оценкой доли полигенной СГХС согласно шкале генетического риска (ШГР) 6-SNP среди пациентов с диагнозом возможная/вероятная/определенная СГХС в Санкт-Петербурге.
Материал и методы. В рамках исследования была проведена апробация гибридной таргетной панели для генодиагностики наследственных дислипидемий, включающей расширенный список генов, ассоциированных с моногенными дислипидемиями, и ряд однонуклеотидных вариантов, связанных с полигенной гиперхолестеринемией и сердечно-сосудистым риском. Генетическое тестирование было выполнено для 125 пациентов.
Результаты. Всего выявлено 26 патогенных и вероятно патогенных вариантов в гене LDLR у 38 пациентов, 6 вариантов неопределенного клинического значения в гене LDLR у 7 пациентов, патогенный вариант в гене APOB c.10580G>A p.Arg3527Gln выявлен у 6 пациентов. Наибольшая выявляемость причинных генетических вариантов (66,7%) была отмечена, как и ожидалось, в подгруппе пациентов с определенной СГХС. В подгруппах с диагнозом возможная и вероятная СГХС преобладали пациенты с высоким полигенным риском развития гиперхолестеринемии.
Заключение. Использование расширенной NGS панели, включающей гены наследственных дислипидемий, а также локусы для расчета ШГР, может быть критически важным для проведения дифференциальной диагностики, определения этиологии гиперхолестеринемии и для персонализации подходов к лечению.
Ключевые слова
Для цитирования:
Мирошникова В.В., Изюмченко А.Д., Музалевская М.В., Легостаева К.В., Грунина М.Н., Драчева К.В., Уразгильдеева С.А., Беркович О.А., Баранова Е.И., Глотов О.С., Куликов А.Н., Гуревич В.С., Пчелина С.Н. Генетическая архитектура семейной гиперхолестеринемии на примере когорты жителей Санкт-Петербурга. Российский кардиологический журнал. 2025;30(10):6432. https://doi.org/10.15829/1560-4071-2025-6432. EDN: MCOSAZ
For citation:
Miroshnikova V.V., Izyumchenko A.D., Muzalevskaya M.V., Legostaeva K.V., Grunina M.N., Dracheva K.V., Urazgildeeva S.A., Berkovich O.A., Baranova E.I., Glotov O.S., Kulikov A.V., Gurevich V.S., Pchelina S.N. Genetic architecture of familial hypercholesterolemia: a cohort of St. Petersburg residents. Russian Journal of Cardiology. 2025;30(10):6432. (In Russ.) https://doi.org/10.15829/1560-4071-2025-6432. EDN: MCOSAZ
Семейная гиперхолестеринемия (СГХС) — распространенное генетическое заболевание с аутосомно-доминантным типом наследования, характеризующееся повышением уровня холестерина липопротеинов низкой плотности (ХС-ЛНП) плазмы крови и высоким риском раннего развития сердечно-сосудистых заболеваний. Распространенность гетерозиготной СГХС в разных регионах России составляет от 1:108 до 1:173 [1]. В то же время 90% случаев СГХС как в мире, так и в России остаются недиагностированными, что подчеркивает необходимость внедрения эффективных программ скрининга СГХС [2].
Моногенные формы СГХС, как правило, обусловлены мутациями в генах рецептора ЛНП — LDLR (90%), аполипопротеина В — APOB (5-10%), пропротеиновой конвертазы субтилизин-кексинового типа 9 — PCSK9 (<1%) [3][4]. Генетическое тестирование СГХС дает возможность провести уточненную стратификацию сердечно-сосудистого риска: наличие патогенных вариантов увеличивает риск ишемической болезни сердца в 22 раза в популяции, а среди лиц с одинаковыми значениями ХС-ЛНП при выявлении патогенных вариантов риск повышается в 3 раза [2]. При этом тип патогенного генетического варианта является независимым предиктором недостижения целевых значений ХС-ЛНП [2]. Вероятность выявления генетической причины заболевания тем выше, чем более выражена клиническая картина (повышение уровня ХС-ЛНП, ксантоматоз, семейный анамнез), и у лиц с диагнозом "определенная СГХС" (>8 баллов по голландским диагностическим критериям) составляет 50-80% [5][6]. Так, в нашем предыдущем исследовании у пациентов с СГХС выявляемость составила 58% [7]. Секвенирование нового поколения (NGS) является основным подходом генетической диагностики СГХС, поскольку позволяет включить в анализ кодирующие области всех генов, при этом кроме канонических генов LDLR, APOB, PCSK9, международным консорциумом в настоящее время рекомендовано исследовать гены фенокопий СГХС (APOE (аполипопротеин Е), LDLRAP1 (адапторный белок для рецептора ЛНП), LIPA (лизосомальная кислая липаза), ABCG5 и ABCG8 (транспортеры стеролов)), для которых существуют отдельные терапевтические подходы [2][4][5]. В частности, нами и другими авторами в России недавно было описано несколько случаев ситостеролемии, связанной с патогенными вариантами в генах ABCG5, ABCG8 и имеющей клиническую картину схожую с СГХС [8].
Важно отметить, что у пациентов с предполагаемым диагнозом СГХС, у которых не удается обнаружить патогенные варианты в вышеперечисленных генах, заболевание может иметь полигенную природу [4]. Потому одним из востребованных подходов к увеличению предсказательной ценности генетического тестирования при СГХС является разработка шкал генетического риска (ШГР) на основе данных исследований по полно-геномному анализу ассоциаций (GWAS), которые могли бы учитывать суммарный вклад рисковых и протективных аллелей в генах, связанных с липидным обменом [9][10]. Таким образом ШГР может использоваться для прогнозирования индивидуального риска развития заболевания [11]. Проведение такого генетического тестирования на текущий момент не входит в клинические рекомендации, однако для полигенной гиперхолестеринемии были предложены различные варианты ШГР [9]. Наиболее часто используемой на данный момент является ШГР 6-SNP, включающая 6 полиморфных генетических вариантов, ассоциированных с уровнем ХС-ЛНП (rs629301 (CELSR2/SORT1), rs1367117 (APOB), rs4299376 (ABCG5/8), rs6511720 (LDLR), rs7412 и rs429358 (APOE)), продемонстрировавшая свою эффективность на нескольких европейских популяциях [12]. Как правило, ШГР помогает объяснить в среднем 20% всех случаев, когда не удается выявить патогенные варианты в генах СГХС [4][5]. В целом, опыт ведущих лабораторий, разрабатывающих решения для диагностики СГХС и других наследственных дислипидемий, показывает целесообразность учета маркеров полигенной СГХС в дизайне таргетной панели [4].
Нами была разработана таргетная панель, включающая расширенный список генов, ассоциированных с моногенными дислипидемиями, и ряд однонуклеотидных вариантов, связанных с полигенной гиперхолестеринемией и сердечно-сосудистым риском по данным GWAS. Цель настоящего исследования — определить частоту патогенных и вероятно патогенных вариантов, ассоциированных с СГХС, на ряду с оценкой доли полигенной СГХС согласно ШГР 6-SNP среди пациентов с диагнозом возможная/вероятная/определенная СГХС в Санкт-Петербурге.
Материал и методы
В исследование было включено 125 пациентов с диагнозом возможная/вероятная/определенная СГХС, которые наблюдались в ПСПбГМУ им. И. П. Павлова или Центре атеросклероза и нарушений липидного обмена на базе ФГБУ "СЗОНКЦ им. Л. Г. Соколова ФМБА" (табл. 1). Диагноз СГХС устанавливался на основании Голландских диагностических критериев [2]. Всем пациентам была проведена генетическая диагностика СГХС. Исследование выполнялось в соответствии со стандартами надлежащей клинической практики (Good Clinical Practice) и принципами Хельсинкской декларации. Исследование было одобрено Локальными этическими комитетами ПСПбГМУ им. И. П. Павлова и СЗОНКЦ им. Л. Г. Соколова. До включения в исследование у участников было получено письменное информированное согласие.
Таблица 1
Характеристики группы пациентов с СГХС
|
Общая выборка |
Подгруппа с возможной СГХС |
Подгруппа с вероятной СГХС |
Подгруппа с определенной СГХС |
|
|
Средний возраст, лет |
51±14 |
51±12 |
55±13 |
49±15 |
|
Пол (М/Ж) |
54/71 |
23/14 |
13/15 |
18/42 |
|
Максимальная концентрация ОХС плазмы крови, ммоль/л |
10,5±2,6 |
9,7±2,5 |
9,9±2,9 |
10,9±2,5 |
|
Концентрация ОХС плазмы крови, ммоль/л |
7,7±2,8 |
7,0±2,4 |
7,4±2,4 |
8,5±3,1 |
|
Концентрация ХС-ЛНП, ммоль/л |
5,3±2,4 |
4,4±1,6 |
5,1±2,2 |
6,2±2,8 |
|
Ксантомы, n (%) |
59 (47%) |
2 (5%) |
9 (32%) |
48 (86%) |
|
Сердечно-сосудистые заболевания, n (%) |
72 (58%) |
19 (51%) |
15 (54%) |
39 (65%) |
Сокращения: ОХС — общий холестерин плазмы крови, СГХС — семейная гиперхолестеринемия, ХС-ЛНП — холестерин липопротеинов низкой плотности.
Генетическая диагностика проводилась в ПСПбГМУ им. И. П. Павлова. ДНК выделяли из лейкоцитов периферической крови фенол-хлороформным методом. Библиотеки готовили с использованием набора реагентов Prep&Seq (Parseq Lab Co, Россия) и панели "Дислипидемия и риск ССЗ", включающей кодирующие регионы следующих генов: ABCA1, ABCG1, ABCG5, ABCG8, ANGPTL3, APOA1, APOA4, APOA5, APOB, APOC2, APOC3, APOE, CETP, CREB3L3, GCK, СYP27A1, CYP7A1, GPD1, GPIHBP1, HNF1A, LCAT, LDLR, LDLRAP1, LIPA, LIPC, LIPG, LMF1, LPL, LRP6, MTTP, MYLIP, NPC1L1, PCSK9, PNPLA5, SAR1B, SCARB1, SORT1, STAP1, TTR, а также полиморфные маркеры rs629301, rs1367117, rs4299376, rs6511720, rs7412, rs429358 (VariFind LM assay IL-v1.1.1, Parseq Lab Co, Россия). Контроль качества библиотек был проведен с помощью Agilent 2100 Bioanalyzer (Agilent, США) и Qubit (Thermo Fisher Scientific, США). Секвенирование проводили в режиме парных ридов 2*150 на секвенаторе MiSeq (Illumina, США). Данные секвенирования были обработаны автоматизированной программой Seq&Go Software (Parseq Lab Co, Россия). Выявленные генетические варианты были проаннотированы и описаны согласно рекомендациям the Human Genome Variation Society (HGVS) (www.hgvs.org). Для оценки клинической релевантности выявленных вариантов нуклеотидных последовательностей использованы базы данных OMIM, gnomAD, ClinVar, HGMD, LOVD и литературные данные. Оценка клинической значимости (патогенности) выявленных вариантов проводилась на основании российских рекомендаций для интерпретации данных, полученных методами массового параллельного секвенирования, а также рекомендаций ACMG2015. Верификация всех выявленных вариантов проводилась методом Сэнгера на секвенаторе Нанофор-05 (Синтол, Россия) с использованием набора реактивов BigDye™ Terminator v3.1 Cycle Sequencing Kit (Applied Biosystems, США), результаты анализировали с помощью программного обеспечения Mutation Surveyor (Soft Genetics, США). Значение ШГР 6-SNP рассчитывалось по взвешенной сумме рисковых аллелей, как описано ранее [12].
Результаты
На рисунке 1 представлены результаты распределения выявленных генетических факторов в зависимости от клинического диагноза пациентов (возможная/вероятная/определенная СГХС). Наибольшая выявляемость причинных генетических вариантов (66,7%) была отмечена, как и ожидалось, в подгруппе пациентов с определенной СГХС. В подгруппе с диагнозом возможная СГХС преобладали пациенты без патогенных генетических вариантов, но с высоким полигенным риском развития гиперхолестеринемии. В качестве критерия высокого полигенного риска принимали значения ≥0,79, что соответствует 80% процентилю, ранее рассчитанному для российской популяции [13]. Средние значения ШГР 6-SNP между исследуемыми группами не различались (0,73 (-0,22-0,93) для возможной, 0,73 (0,21-1,10) для вероятной, 0,73 (0,15-1,00) для определенной СГХС, p>0,05).
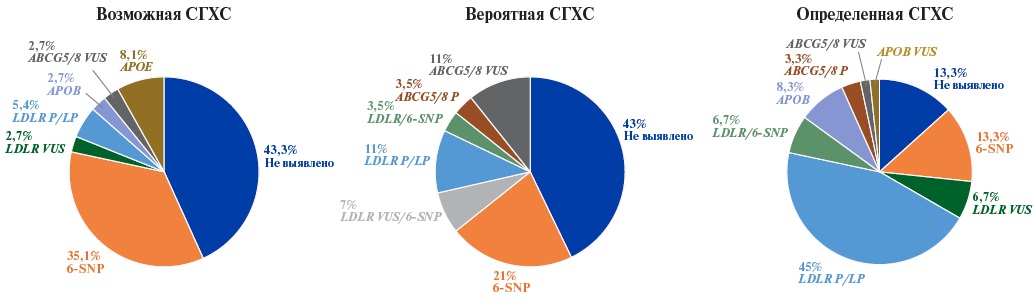
Рис. 1. Распределение генетических вариантов и доля пациентов с высоким полигенным риском в зависимости от клинического диагноза СГХС.
Сокращения: СГХС — семейная гиперхолестеринемия, P — патогенный, LP — вероятно патогенный, VUS — вариант неопределенного клинического значения, 6-SNP — высокий полигенный риск по ШГР 6-SNP.
Примечание: цветное изображение доступно в электронной версии журнала.
Список всех выявленных патогенных/вероятно патогенных вариантов, а также вариантов неопределенного клинического значения, представлен в таблице 2. Чаще всего у пациентов с СГХС выявляли варианты в гене LDLR (82,6% от всех выявленных патогенных и вероятно патогенных вариантов), в т.ч. в настоящей работе были обнаружены новые не описанные ранее варианты (табл. 2). Наиболее частый патогенный вариант в гене APOB c.10580G>A p.Arg3527Gln был выявлен у 6 пациентов (5%). Дисбеталипопротеинемия выявлена у 3 пациентов. Также было выявлено 4 гетерозиготных носителя патогенных вариантов (один из них носитель генотипа e2e2 гена APOE) и 5 носителей вариантов неопределенного клинического значения в генах ABCG5/ABCG8 (табл. 2). У одного из пациентов без патогенных вариантов в основных генах СГХС были выявлены 2 редких варианта в гене LDLRAP1: rs386629678 chr1:25563141_25563142delinsCA c.604_605delinsCA p.Ser202His, частота неизвестна; rs147242385 chr1:25566914C>T c.849C>T p.Tyr283=, частота 0,000055).
Таблица 2
Список выявленных вариантов в генах LDLR, APOB, ABCG5/8
|
Количество пациентов |
Геномная координата (GRCh38) |
Замена в транскрипте и в белке |
dbSNP ID |
Clinvar ID |
Патогенность |
|
Варианты в гене LDLR |
|||||
|
1 |
chr19: 11100252 |
c.97C>T p.Gln33Ter |
rs121908024 |
3683 |
патогенный |
|
1 |
chr19: 11100255 |
c.100T>G p.Cys34Gly |
rs879254405 |
251018 |
патогенный |
|
1 |
chr19: 11100347 |
c.190+2T>C |
новый |
– |
неопределенного значения |
|
1 |
chr19: 11102732 |
c.259T>G p.Trp87Gly |
rs121908025 |
3685 |
патогенный |
|
1 |
chr19: 11102774 |
c.301G>A p.Glu101Lys |
rs144172724 |
161266 |
патогенный |
|
1 |
chr19: 11102787 |
c.313+1G>A |
rs112029328 |
3736 |
патогенный |
|
1 |
chr19: 11102788 |
c.313+2T>G |
новый |
– |
неопределенного значения |
|
1 |
сhr19: 11105334 |
c.428G>T p.Cys143Phe |
rs879254522 |
523723 |
вероятно патогенный |
|
1 |
сhr19: 11105337 |
c.433_434dupG (p.Val145Glyfs*35) |
rs2077271255 |
870320 |
патогенный |
|
1 |
сhr19: 11105439 |
c.533A>C p.Asp178Ala |
новый |
– |
вероятно патогенный |
|
2 |
chr19: 11105557 |
c.651delTGG p.Gly219del |
rs121908027 |
226329 |
патогенный |
|
1 |
chr19: 11106638 |
c.768C>A p.Asp256Glu |
rs879254671 |
438322 |
вероятно патогенный |
|
1 |
chr19: 11106666 |
c.796G>A p.Asp266Asn |
rs875989907 |
226334 |
патогенный |
|
1 |
chr19: 11106668 |
c.798T>A p.Asp266Glu |
rs139043155 |
161287 |
патогенный |
|
1 |
chr19: 11106680 |
c.810C>A p.Cys270Ter |
rs773328511 |
251465 |
патогенный |
|
1 |
chr19: 11107461 |
c.887G>A p.Cys296Tyr |
rs879254707 |
251502 |
вероятно патогенный |
|
1 |
chr19: 11107462 |
c.888C>A p.Cys296Ter |
rs879254708 |
251504 |
патогенный |
|
4 |
chr19: 11110697 |
c.986G>A p.Cys329Tyr |
rs761954844 |
226344 |
патогенный |
|
3 |
chr19: 11110759 |
c.1048C>T p.Arg350Ter |
rs769737896 |
226342 |
патогенный |
|
1 |
сhr19: 11111639 |
c.1186G>C p.Gly396Arg |
rs879254820 |
870321 |
неопределенного значения |
|
1 |
chr19: 11113268 |
c.1187-10G>A |
rs765696008 |
588170 |
патогенный |
|
4 |
сhr19: 11113293 |
c.1202T>A p.Leu401His |
rs121908038 |
3735 |
вероятно патогенный |
|
1 |
chr19: 11113542 |
c.1366C>T p.Leu456Phe |
rs761004553 |
926526 |
вероятно патогенный* |
|
1 |
сhr19: 11113652-11113653 |
c.1478_1479delCT p.(Ser493Cysfs*42) |
rs869025453 |
222688 |
патогенный |
|
1 |
chr19: 11116873 |
c.1720C>T p.Arg574Cys |
rs185098634 |
183123 |
неопределенного значения |
|
1 |
chr19: 11116886 |
c.1732G>T p.Val578Phe |
rs1301458707 |
1321219 |
неопределенного значения |
|
3 |
chr19: 11116928 |
c.1775G>A p.Gly592Glu |
rs137929307 |
161271 |
вероятно патогенный |
|
1 |
chr19: 11120412 |
c.2030G>A p.Cys677Tyr |
rs875989938 |
252178 |
вероятно патогенный |
|
1 |
chr19: 11120436 |
c.2054C>T p.Pro685Leu |
rs28942084 |
3702 |
патогенный |
|
1 |
chr19: 11123263 |
c.2230C>T p.Arg744Ter |
rs200793488 |
430795 |
патогенный |
|
1 |
chr19: 11128085 |
c.2389G>A p.Val797Met |
rs750518671 |
226393 |
вероятно патогенный |
|
2 |
chr19: 11128090 |
c.2389+5G>C |
rs879255191 |
661713 |
неопределенного значения |
|
1 |
chr19: 11129518 |
c.2395del p.Leu799SerfsTer130 |
новый |
– |
патогенный |
|
Варианты в гене APOB |
|||||
|
6 |
chr2: 21006288 |
c.10580G>A p.Arg3527Gln |
rs5742904 |
17890 |
вероятно патогенный |
|
1 |
chr2: 21001940- 21001942 |
c.13477delCAG p.Gln4494del |
rs562574661 |
265896 |
неопределенного значения |
|
Варианты в гене ABCG5 |
|||||
|
1 |
chr2: 43822924 |
c.1336C>T p.Arg446Ter |
rs199689137 |
30485 |
патогенный |
|
1 |
chr2: 43828024 |
c.593G>A p.Arg198Gln |
rs141828689 |
284636 |
неопределенного значения |
|
Варианты в гене ABCG8 |
|||||
|
3 |
chr2: 43872094 |
c.1083G>A p.Trp361Ter |
rs137852987 |
4967 |
патогенный |
|
1 |
chr2: 43852376 |
c.584T>A p.Leu195Gln |
rs1167870780 |
2203056 |
неопределенного значения |
|
1 |
chr2: 43875362 |
c.1705T>C p.Ser569Pro |
rs776335488 |
2739113 |
неопределенного значения |
|
1 |
chr2: 43877641 |
c.1837T>C p.Tyr613His |
rs189249032 |
288712 |
неопределенного значения |
|
1 |
chr2: 43877815 |
c.1924G>A p.Ala642Thr |
rs113005049 |
288467 |
неопределенного значения |
Примечание: * — данный вариант можно считать вероятно патогенным (PM1 PM5 PP1 PP3).
Обсуждение
Генетическая диагностика СГХС в настоящее время представляет определенные проблемы, т.к. не у всех пациентов с гиперхолестеринемией и ранним развитием сердечно-сосудистых заболеваний удается установить причину заболевания. Увеличению ценности генетического тестирования должно способствовать использование расширенных панелей для дифференциальной диагностики других дислипидемий, сходных по клиническим проявлениям с СГХС, развитие подходов к оценке полигенного риска заболевания, а также обследование семей и реклассификация вариантов неизвестного клинического значения.
В настоящей работе мы показываем эффективность применения новой диагностической NGS панели, которая позволяет проводить не только генодиагностику моногенной дислипидемии, включая рекомендованные гены, ассоциированные с СГХС и ее фенокопиями [2][4], но и вычислить значение наиболее используемой сегодня ШГР 6-SNP на основании носительства шести вариантов в генах липидного обмена LDLR, APOB, APOE, ABCG5/8 и SORT1 [5][6][11].
Ранее при использовании предложенной NGS панели для дифференциальной диагностики дислипидемий, нами был описан клинический случай ситостеролемии, обусловленной биаллельным носительством патогенных вариантов в гене ABCG8 [8]. В настоящем исследовании мы выявили четырех носителей патогенных вариантов в генах ABCG5 и ABCG8. Следует отметить, что вклад гетерозиготного носительства патогенных вариантов в генах ABCG5 и ABCG8 в развитие СГХС остается дискутабельным. Тем не менее варианты в этих генах могут предрасполагать к развитию полигенной СГХС, и, основываясь на результатах GWAS исследований, полиморфный вариант rs4299376 ABCG5/ABCG8 был включен в ШГР 6-SNP. Транспортеры стеролов ABCG5 и ABCG8 с тканеспецифичной экспрессией в печени и кишечнике участвуют в регуляции всасывания холестерина в кишечнике и выведении его через желчь. Среди атерогенных дислипидемий можно также выделить семейную дисбеталипопротеинемию, частота которой, вероятно, недооценена и по последним данным составляет 0,5-0,8% (1 на 118-184) в европейском регионе России [13]. В нашем исследовании выявлены 2 пациента с генотипом e2e2 и один пациент с вариантом p.Arg154Cys в гене APOE. Выявление таких вариантов позволило своевременно инициировать патогенетическое лечение пациентов с использованием эзетимиба в случае ситостеролемии, комбинации статинов и фенофибрата — для пациентов с дисбеталипопротеинемией.
В нашем исследовании варианты в канонических сайтах сплайсинга составили 3,4%, в основном все они были ранее описаны у российских пациентов с СГХС [3][14]. Выявленный нами новый вариант c.190+2T>C расположен в донорном сайте интрона 2, ранее описанные варианты в данном сайте c.190+1G>A, c.190+2T>G и 190+4A>T классифицированы как вероятно патогенные. Патогенные и вероятно патогенные варианты нарушения сплайсинга мРНК могут составлять по разным оценкам от 9% до 25% от всех мутаций, однако для доказательства их патогенности требуются дополнительные исследования [14].
В целом ~47% вариантов классифицируются как варианты неопределенного клинического значения, из-за отсутствия достаточных доказательств их связи с заболеванием/фенотипом, отсутствия функциональных исследований, а некоторые варианты были описаны только единожды [2]. Реклассификация вариантов неопределенного клинического значения имеет важное значение. Как показывает практика, в случае генов APOB и PCSK9 такие варианты в большинстве случаев не влияют на функцию рецептора ЛНП и маловероятно вызывают заболевание. В то же время ~50% вариантов неопределенного клинического значения в гене LDLR впоследствии переклассифицируются в патогенные и вероятно патогенные при появлении новых данных [5]. В настоящее время для оценки патогенности также можно воспользоваться данными, полученными в результате моделирования молекулярной структуры белка рецептора ЛНП [15]. Так, например, нами выявлен семейный случай СГХС, ассоциированный с вариантом неопределенного клинического значения c.1366C>T p.Leu456Phe в гене LDLR. По данным моделирования in silico данная аминокислотная замена приводит к нарушению структуры и функции рецептора ЛНП [15].
В настоящем исследовании патогенные и вероятно патогенные варианты закономерно кластеризуются в подгруппе пациентов с определенной СГХС (рис. 1), для которых характерно более значимое повышение уровня ХС-ЛНП и более выраженная клиническая картина, а пациенты с высоким значением полигенного риска преобладают в подгруппе в возможной СГХС, предполагая больший вклад полигенного компонента. Аналогичные результаты были получены в работе Rieck L, et al., основным драйвером развития заболевания у пациентов с определенной СГХС являются патогенные и вероятно патогенные генетические варианты [6]. Несмотря на то, что полигенный фон может модулировать пенетрантность моногенных вариантов, в этой области требуются дальнейшие исследования [2][12]. Следует отметить, что предложенные в настоящее время ШГР обладают ограниченной клинической эффективностью по следующим причинам: 1) небольшой размер первоначальных исследований GWAS, что повлияло на точность оценки влияния отдельных вариантов на риск заболевания; 2) ограниченные вычислительные методы для создания ШГР; 3) отсутствие больших наборов данных, необходимых для тестирования и валидации ШГР; 4) необходимость учитывать популяционные особенности [9][10]. По результатам исследований, проведенных в последнее время, рядом авторов было высказано предположение, что редкие рисковые аллели кластеризуются внутри семей, однако в настоящее время такие данные немногочисленны ввиду сложности получения биоматериала в обширных родословных [6][10]. Таким образом, будущие исследования должны оценить прогностическую значимость различных ШГР, учитывающих как частые полиморфные варианты, так и редкие варианты, для оценки риска развития дислипидемии и сердечно-сосудистых осложнений.
Ограничения исследования. Небольшой размер выборки и, поскольку пациенты набирались в специализированных центрах, превалирование в выборке пациентов с диагнозом "определенная СГХС", использование ШГР с небольшим набором вариантов нуклеотидной последовательности.
Заключение
Использование расширенной NGS панели, включающей гены наследственных дислипидемий, а также локусы для расчета ШГР, может быть критически важным как для проведения дифференциальной диагностики, так и для выявления групп с полигенной природой гиперхолестеринемии. Данный подход имеет ключевое значение для определения этиологии гиперхолестеринемии, а кроме того, может быть использован для персонализации подходов к лечению, т.е. для выбора препаратов и усиленного контроля дозировок, а также как основание для ранней профилактики.
Следует, однако, отметить, что в настоящее время полигенная оценка риска пока ещё не является надежным инструментом клинической практики. Дальнейшие исследования позволят предложить использование ШГР для скрининга общего риска сердечно-сосудистых заболеваний и, возможно, предоставят дополнительную информацию для пациентов с высоким риском с известной моногенной причиной.
Отношения и деятельность: все авторы заявляют об отсутствии потенциального конфликта интересов, требующего раскрытия в данной статье.
Список литературы
1. Meshkov AN, Ershova AI, Kiseleva AV, et al. The prevalence of heterozygous familial hypercholesterolemia in selected regions of the Russian Federation: The FH-ESSE-RF study. Journal of Personalized Medicine. 2021;11(6):464. doi:10.3390/jpm11060464.
2. Sturm AC, Knowles JW, Gidding SS, et al. Clinical genetic testing for familial hypercholesterolemia: JACC scientific expert panel. Journal of the American College of Cardiology. 2018;72(6):662-80. doi:10.1016/j.jacc.2018.05.044.
3. Meshkov A, Ershova A, Kiseleva A, et al. The LDLR, APOB, and PCSK9 variants of index patients with familial hypercholesterolemia in Russia. Genes. 2021;12(1):66. doi:10.3390/genes12010066.
4. Мирошникова В.В., Пчелина С.Н., Донников М.Ю. и др. Генетическое тестирование в кардиологии с помощью NGS панели: от оценки риска заболевания до фармакогенетики. Фармакогенетика и фармакогеномика. 2023;(1):7-19. doi:10.37489/2588-0527-2023-1-7-19.
5. Medeiros AM, Alves AC, Miranda B, et al. Unraveling the genetic background of individuals with a clinical familial hypercholesterolemia phenotype. Journal of Lipid Research. 2024;65(2):100490. doi:10.1016/j.jlr.2023.100490.
6. Rieck L, Bardey F, Grenkowitz T, et al. Mutation spectrum and polygenic score in German patients with familial hypercholesterolemia. Clinical genetics. 2020;98(5):457-67. doi:10.1111/cge.13826.
7. Miroshnikova VV, Romanova OV, Ivanova ON, et al. Identification of novel variants in the LDLR gene in Russian patients with familial hypercholesterolemia using targeted sequencing. Biomedical Reports. 2021;14(1):15. doi:10.3892/br.2020.1391.
8. Miroshnikova VV, Vasiluev PA, Linkova SV, et al. Pediatric Patients with Sitosterolemia: Next-Generation Sequencing and Biochemical Examination in Clinical Practice. Journal of Personalized Medicine. 2023;13(10):1492. doi:10.3390/jpm13101492.
9. Бойцов С.А., Погосова Н.В., Аншелес А.А. и др. Кардиоваскулярная профилактика 2022. Российские национальные рекомендации. Российский кардиологический журнал. 2023;28(5):5452. doi:10.15829/1560-4071-2023-5452. EDN: EUDWYG.
10. Khera AV, Chaffin M, Aragamet KG, et al. Genome-wide polygenic scores for common diseases identify individuals with risk equivalent to monogenic mutations. Nature genetics. 2018;50(9):1219-24. doi:10.1038/s41588-018-0183-z.
11. Мешков А.Н., Киселева А.В., Ершова А.И. и др. Варианты генов ANGPTL3, ANGPTL4, APOA5, APOB, APOC2, APOC3, LDLR, PCSK9, LPL и риск ишемической болезни сердца. Российский кардиологический журнал. 2022;27(10):5232. doi:10.15829/1560-4071-2022-5232. EDN: FAKGMM.
12. Futema M, Shah S, Cooper JA, et al. Refinement of variant selection for the LDL cholesterol genetic risk score in the diagnosis of the polygenic form of clinical familial hypercholesterolemia and replication in samples from 6 countries. Clinical chemistry. 2015;61(1):231- 8. doi:10.1373/clinchem.2014.231365.
13. Blokhina AV, Ershova AI, Kiseleva AV, et al. Clinical and biochemical features of atherogenic hyperlipidemias with different genetic basis: A comprehensive comparative study. Plos one. 2024;19(12): e0315693. doi:10.1371/journal.pone.0315693.
14. Vasilyev V, Zakharova F, Bogoslovskay T, Mandelshtam M. Familial hypercholesterolemia in Russia: three decades of genetic studies. Frontiers in genetics. 2020;11:550591. doi:10.3389/fgene.2020.550591.
15. Guo J, Gao Y, Li X, et al. Systematic prediction of familial hypercholesterolemia caused by low-density lipoprotein receptor missense mutations. Atherosclerosis. 2019;281:1-8. doi:10.1016/j.atherosclerosis.2018.12.003.
Об авторах
В. В. МирошниковаРоссия
Мирошникова Валентина Вадимовна — к.б.н., Петербургский институт ядерной физики им. Б.П. Константинова НИЦ «Курчатовский институт», с.н.с. Лаборатории молекулярной генетики человека Отделения молекулярной и радиационной биофизики; Первый Санкт-Петербургский государственный медицинский университет им. акад. И.П. Павлова, зав. Лабораторией медицинской генетики Отдела молекулярно-генетических и нанобиологических технологий
мкр. Орлова роща, д.1, Гатчина, 188300,
ул. Льва Толстого, д.6-8, Санкт-Петербург, 197022
А. Д. Изюмченко
Россия
Изюмченко Артем Дмитриевич — аспирант отдела молекулярно-генетических и нанобиологических технологий; лаборант-исследователь лаборатории молекулярной генетики человека отделения молекулярной и радиационной биофизики
мкр. Орлова роща, д.1, Гатчина, 188300,
ул. Льва Толстого, д.6-8, Санкт-Петербург, 197022
М. В. Музалевская
Россия
Музалевская Мария Вячеславовна — врач-кардиолог; аспирант
проспект Культуры, д.4, Санкт-Петербург,
проспект Динамо, д.3, Санкт-Петербург, 197110
К. В. Легостаева
Россия
Легостаева Кристина Владимировна — ассистент кафедры пропедевтики внутренних болезней с клиникой им. акад. М. Д. Тушинского, врач-терапевт терапевтического отделения № 3
ул. Льва Толстого, д.6-8, Санкт-Петербург, 197022
М. Н. Грунина
Россия
Грунина Мария Николаевна — к.б.н., Петербургский институт ядерной физики им. Б.П. Константинова НИЦ «Курчатовский институт», м.н.с. Лаборатории молекулярной генетики человека Отделения молекулярной и радиационной биофизики; Первый Санкт-Петербургский государственный медицинский университет им. акад. И.П. Павлова, м.н.с. Отдела молекулярно-генетических и нанобиологических технологий
мкр. Орлова роща, д.1, Гатчина, 188300,
ул. Льва Толстого, д.6-8, Санкт-Петербург, 197022
К. В. Драчева
Россия
Драчева Ксения Владимировна — к.б.н., м.н.с. отдела молекулярно-генетических и нанобиологических технологий
мкр. Орлова роща, д.1, Гатчина, 188300
С. А. Уразгильдеева
Россия
Уразгильдеева Сорейя Асафовна — д.м.н., Научно-клинический и образовательный центр «Кардиология» Санкт-Петербургского государственного университета, ведущий научный сотрудник Научно-клинического отдела атеросклероза (Центр атеросклероза и нарушений липидного обмена); консультант-кардиолог Центра атеросклероза и нарушений липидного обмена СЗОНКЦ им. Л.Г. Соколова; руководитель центра липидологии курортной кардиологической клиники «Чёрная речка»
проспект Культуры, д.4, Санкт-Петербург,
проспект Динамо, д.3, Санкт-Петербург, 197110,
Приморское шоссе, д.648, пос. Молодежное, Санкт-Петербург, 197729
О. А. Беркович
Россия
Беркович Ольга Александровна — д.м.н., профессор, профессор кафедры терапии факультетской с курсом кардиологии, эндокринологии и функциональной диагностики с клиникой им. акад. Г. Ф. Ланга, зав. лабораторией ишемической болезни сердца института сердечно-сосудистых заболеваний
ул. Льва Толстого, д.6-8, Санкт-Петербург, 197022
Е. И. Баранова
Россия
Баранова Елена Ивановна — д.м.н.; профессор кафедры терапии факультетской с курсом кардиологии, эндокринологии и функциональной диагностики с клиникой им. акад. Г. Ф. Ланга
ул. Льва Толстого, д.6-8, Санкт-Петербург, 197022
О. С. Глотов
Россия
Глотов Олег Сергеевич — д.б.н., зав. отделом экспериментальной медицинской вирусологии, молекулярной генетики и биобанкинга
ул. Профессора Попова, д.9, литера А, Санкт-Петербург, 197022
А. Н. Куликов
Россия
Куликов Александр Николаевич — д.м.н.; Первый Санкт-Петербургский государственный медицинский университет им. акад. И.П. Павлова, заведующий кафедры пропедевтики внутренних болезней с клиникой имени академика М.Д.Тушинского, заведующий кафедры функциональной диагностики, директор научно-клинического исследовательского центра
ул. Льва Толстого, д.6-8, Санкт-Петербург, 197022
В. С. Гуревич
Россия
Гуревич Виктор Савельевич — д.м.н., профессор; Научно-клинический и образовательный центр «Кардиология» Санкт-Петербургского государственного университета, руководитель Научно-клинического отдела атеросклероза; профессор кафедры госпитальной терапии СЗГМУ им. И.И. Мечникова, руководитель Центра атеросклероза и нарушений липидного обмена СЗОНКЦ им. Л.Г. Соколова, вице-президент Национального Общества по изучению Атеросклероза
проспект Культуры, д.4, Санкт-Петербург,
проспект Динамо, д.3, Санкт-Петербург, 197110,
, ул. Кирочная, д.41, г. Санкт-Петербург, 191015
С. Н. Пчелина
Россия
Пчелина Софья Николаевна — д.б.н.; Петербургский институт ядерной физики им. Б.П. Константинова НИЦ «Курчатовский институт», зав. Лабораторией молекулярной генетики человека Отделения молекулярной и радиационной биофизики; Первый Санкт-Петербургский государственный медицинский университет им. акад. И.П. Павлова, руководитель Отдела молекулярно-генетических и нанобиологических технологий
мкр. Орлова роща, д.1, Гатчина, 188300,
ул. Льва Толстого, д.6-8, Санкт-Петербург, 197022
Дополнительные файлы
- У пациентов с возможной и вероятной семейной гиперхолестеринемией существенный вклад вносит полигенная гиперхолестеринемия (до 35,1% для примененной шкалы генетического риска (ШГР)).
- Расширенная NGS панель, включающая гены наследственных дислипидемий, а также локусы для расчета ШГР, может эффективно применяться для проведения дифференциальной диагностики, определения этиологии гиперхолестеринемии и для персонализации подходов к лечению.
Рецензия
Для цитирования:
Мирошникова В.В., Изюмченко А.Д., Музалевская М.В., Легостаева К.В., Грунина М.Н., Драчева К.В., Уразгильдеева С.А., Беркович О.А., Баранова Е.И., Глотов О.С., Куликов А.Н., Гуревич В.С., Пчелина С.Н. Генетическая архитектура семейной гиперхолестеринемии на примере когорты жителей Санкт-Петербурга. Российский кардиологический журнал. 2025;30(10):6432. https://doi.org/10.15829/1560-4071-2025-6432. EDN: MCOSAZ
For citation:
Miroshnikova V.V., Izyumchenko A.D., Muzalevskaya M.V., Legostaeva K.V., Grunina M.N., Dracheva K.V., Urazgildeeva S.A., Berkovich O.A., Baranova E.I., Glotov O.S., Kulikov A.V., Gurevich V.S., Pchelina S.N. Genetic architecture of familial hypercholesterolemia: a cohort of St. Petersburg residents. Russian Journal of Cardiology. 2025;30(10):6432. (In Russ.) https://doi.org/10.15829/1560-4071-2025-6432. EDN: MCOSAZ

















































